发布者:林章 发布时间:2017-10-09 动态浏览次数:7976

欧阳松应 博士,二级教授,博士、硕士研究生导师。福建师范大学副校长、生命科学学院院长,南方生物医学研究中心主任。国家杰出青年基金获得者、享受国务院政府特殊津贴人员、福建省百人计划获得者、福建省百千万人才工程人选,“闽江学者”特聘教授。
中央财政支持地方高校发展专项资金项目带头人,国家级生物学实验教学示范中心主任,病原微生物感染机理与干预福建省高校重点实验室主任,“福建省现代微生物与免疫学创新平台”带头人,福建省生物学“双一流”建设高原学科带头人,福建省生物与医药专业学位导师团队带头人,“生物学”一级学科博士点学术带头人。福建省“百人计划”获得者、福建省百千万人才工程人选、“闽江学者”特聘教授、福建省团队科技特派员带头人、北京市科技新星、福建省人力资源和社会保障厅2018年第一批省直中直单位引进创业创新领军人才(B类引进高层次人才)、中国科学院青年创新促进会会员、中国科学院卢嘉锡青年人才奖获得者、福建省“高校新世纪优秀人才支持计划”入选者。入选2021年福建师范大学优秀研究生指导教师。
中国生物物理学会理事及分子生物物理分会二届理事,中国微生物学会理事及微生物学与免疫学专业委员会委员、病毒学专业委员会委员,中国生物化学与分子生物学会理事及酶学专业分会委员,中国病理生理学会蛋白质修饰与疾病专业委员会委员,中国免疫学会自身免疫分会常委,福建省微生物学会副理事长,福建省药理学会常务理事。International Advisory Board of 《Small Structures》(IF:15.9),Associate Editor in 《Frontiers in Microbiology - Phage Biology》(IF:6.064),Review Editor in《Frontiers in Molecular Biosciences - Structural Biology》(IF:6.113),《高校生物学教学研究(电子版)》常务编委、《中国细胞生物学学报》编委,《福建师范大学学报》编委。
主要研究方向:病原微生物感染致病机理及干预。
办公电话:0591-22868199(Office),0591-22868072(Lab)
电子邮件(E-mail): ouyangsy@@fjnu.edu.cn(请删除一个@以防垃圾邮件)
通讯地址:福建福州闽侯区福建师范大学旗山校区理工楼11#南方生物医学研究中心412室(邮编:350117)
一、简历 & 研究工作摘要
1997.07-2001.09 南昌大学 生物科学工程系 学士
2001.08-2004.07 吉林大学和平校区(原解放军军需大学) 生物化学与分子生物学教研室硕士
2004.08-2007.06 北京协和医学院(清华大学)(原中国协和医科大学)基础医学院生物化学与分子生物学系博士
2007.07-2010.08 中国科学院生物物理研究所 助理研究员
2010.09-2013.11 中国科学院生物物理研究所 副研究员
2013.12-2017.05 中国科学院生物物理研究所研究员
2015.12-2017.05 中国科学院生物物理研究所 博士生导师
2017.06- 至今 中国科学院生物物理研究所 客座研究员
2015.09-2016.09 美国UCLA微生物免疫分子遗传系 高级访问学者
2017.06- 至今 福建师范大学生命科学学院/南方生物医学研究中心/福建省天然免疫生物学重点实验室/细胞逆境响应与代谢调控福建省高校重点实验室 教授、博导
在Nature, Nat Immunol, Nat Cell Biol, Immunity, Nat Commun(2篇), PNAS(2篇), Cell Res(2篇), EMBO J(2篇), PLoS Biology等国际知名杂志发表SCI学术论文90余篇,其中通讯或第一作者(含共同)共60余篇。所发表论文总引用次数超过4400余次,单篇最高引用超过430次,引用次数超过100的有9篇,h-index 34。已获得软件著作权2部,申请专利18项,共同主编学术专著1部、参编学术著作4部。主持或承担科技部、国家自然基金委、中国科学院、北京市科委、福建省教育厅等科研和人才项目40项。科研成果曾与我国“大亚湾中微子实验发现新的中微子振荡模式”和“iPSC研究新发现”等当年具有重要影响力的科研成果并列被中国科学院《2013科学发展报告》收录为“中国科学家代表性成果”,向全国人大、全国政协会议代表及国家领导人和有关部门领导提供,供高层科学决策参考,并在社会公开发行;被《中国科学院重大成果年报(2012)》收录,入选《中国科学院国际科技合作年度述评》2012年度报告的《国际科技合作重要进展》。
曾为Cell Res , EMBO J, Nucleic Acids Research, Cancer Research, PNAS, Plant Biotechnology Journal , Acta Cryst D, Journal of Molecular Cell Biology, Trends in Microbiology, Bioinformatics, Theranostics, Oncotarget, J Virol, Protein Cell, Scientific Reports, J Gen Virol, Science China: Life Science, 生物化学与生物物理进展,科学通报等杂志审稿人和国内国际基金项目评审人(如捷克科学基金会(Czech Science Foundation)项目评审人)、北京市自然基金会评专家。先后受邀做国际国内学术会议报告60余场次。
二、研究方向
本实验室受国家重点研发计划课题、国家自然科学基金、福建省相关厅局及企业资助,具备良好的的硬件条件、一流的科研平台和充足的科研经费,主要研究方向:运用X射线晶体学、冷冻电镜和其它生物物理、细胞生物学方法研究病原体与宿主天然免疫系统间相互作用的分子机制。
1. 病毒感染、转录复制机制
RNA病毒是引起人、畜、禽类、水产经济类动物重大传染性疾病的主要病原体。近年来,随着埃博拉病毒、寨卡病毒、中东呼吸道综合征冠状病毒、潜在大流行H5N1禽流感病毒等新发再发RNA病毒的出现,病毒感染性疾病带来的生物安全威胁日益受到广泛重视,已经成为公共卫生领域面临的全球性挑战。同时,新发再发病毒研究事关国家生物安全核心利益,一旦发生必须尽快为国家防控方案提供参考,对基于结构生物学方法解析这些病原体蛋白功能位点要求较高,既需要迅速反应、尽早获得可靠结论,还要求充分结合结构生物学和细胞生物学、病毒学、生物化学、合成生物学、抗体研发等交叉学科技术为潜在治疗药物研究打下基础。
本课题组主要针对RNA病毒中具有代表性的布尼亚病毒等新发再发病毒为主要研究对象,今后的工作将继续夯实当前研究基础,以结构生物学和细胞生物学技术为主线,解析RNA病毒转录复制复合体的晶体结构,阐明病毒转录复制的分子机制,着重分析新发传染病病原体蛋白功能位点,同时阐明病毒转录复制调控及其与宿主相互作用的分子机制,发现RNA病毒转录复制机制的异同,探索干预RNA病毒转录复制的新机制和新手段,进一步拓展相关基础研究成果在生物医学和国防安全领域的应用。解析病毒转录复制及其与宿主相互作用复合体的精确三维结构,对于揭示病毒转录复制的分子机制和发现抗病毒新靶标具有关键的意义,既契合国家传染病防控的重大需求,也是当前蛋白质科学研究的国际前沿和热点。
2. 重要致病性病原菌与宿主相互作用分子机制的研究
紧密围绕细菌性传染病防控和国家安全(生物反恐)等国家重大需求,以重要胞内病原菌为研究对象,如以致病性嗜肺军团菌(Legionella pneumophila)、结核杆菌(Mycobacterium tuberculosis)、弗朗西斯菌(Francisella tularensis)等为模型,系统地筛选发现与病原菌入侵以及胞内存活相关的病原菌致病因子和宿主因子,在分子水平上研究病原菌蛋白与宿主蛋白之间的相互作用,尤其是发现病原菌与宿主互作中的新的作用机制,泛素及类泛素化蛋白等修饰及其它新型修饰如何调控病原菌进入宿主细胞并在胞内存活及相关信号通路的分子机制,深入揭示了其发生作用的分子机制,为新型药物和疫苗的设计提供了理论基础。
主要研究领域包括:1)与病原菌感染致病相关的关键宿主因子的鉴定及机制研究;2)病原菌致病因子的鉴定及与宿主蛋白相互作用的调控机制研究,如病原菌效应蛋白抑制宿主免疫通路分子机制,结核杆菌分泌蛋白调控宿主免疫应答机制研究等;3)致病性细菌蛋白抑制宿主的免疫信号通路新机制。
3. 宿主天然免疫反应的分子机制
真核生物的天然免疫(又称固有免疫或非特异性免疫)反应(innate immune response),发生于系统发育的早期和抗感染应答的初期阶段,是机体抵抗病原微生物入侵、保护自身的第一道屏障。天然免疫系统一方面可以通过炎症反应等机制直接清除感染,另一方面还可以诱导获得性免疫(acquired immunity)系统的活化,进而激发更加高效和特异性的免疫应答,故天然免疫是免疫之本。天然免疫系统通过机体的模式识别受体(pattern-recognition receptors, PRRs)对入侵微生物中高度保守的“分子标签”(molecular signature)——病原相关分子模式(pathogen-associated molecular patterns,PAMPs)进行特异性识别,从而感知病毒、细菌等外来微生物入侵并激活不同的胞内信号通路和基因表达,启动机体免疫反应。天然免疫如同机体的眼睛,监视与识别各种不同的疾病相关分子模式。因此,了解机体免疫系统对外源核酸、蛋白质以及分泌的小分子等的识别和检测的分子基础是天然免疫反应研究的重要内容。
总之,本课题组综合运用X-射线晶体学方法、Cryo-EM、生化与分子生物学、细胞生物学等技术致力于阐明病原体,如病毒/噬菌体、致病菌感染与宿主天然免疫应答中胞内DNA感受因子识别、激活STING引起I型IFN产生、病原-宿主相互作用等生命过程中关键科学问题。
三、指导硕、博研究生及博士后
本课题组长期招收“071010生物化学与分子生物学”专业博士、硕士研究生,“071009细胞生物学”及“086000生物与医药(专业学位)”硕士研究生,同时高薪吸纳相关专业博士后人员。也欢迎有志从事科学研究工作的本科生到实验室交流学习。
硕士
硕士研究生国家奖学金每生每年20000元;国家助学金每生每年6000元;学业奖学金:一、二、三等奖每生每年分别为10000元、6000元、3000元;“三助”中的“助教”、“助管”岗位津贴每生每月不低于500元,助研津贴由导师或课题组发放。此外,学校或学院对本校研究生推免或考取本校研究生给予奖励。
学校开展优秀科研成果、优秀学位论文等项目评选活动,鼓励研究生参加高水平学术会议和到高水平大学、科研院所访学。同时,积极引入多种社会资金,对研究生的学习、生活进行奖励或资助。录取类别为定向就业的学生不享受国家奖学金、国家助学金。
福建省研究生优秀学位论文:2016级研究生叶维伟,2017级研究生叶阳苗
研究生学位论文培育项目资助:周彪、余婷、陈静、王嘉君、王婧婷
国家奖学金:叶阳苗、许小龙、余婷、罗纪维等
2019年“十佳学术海报论文”获奖:2018级研究生许小龙
第三届全国病毒学青年学者学术研讨会上获得Poster奖(二等):2016级硕士研究生叶维伟
四、近期主要工作和进展
1. 罗招庆/欧阳松应合作发表《Nature》解析一种谷氨酸化修饰的新机制(附:专家点评)
2019年7月22日,Nature杂志在线发表了由美国普渡大学罗招庆教授和福建师范大学欧阳松应教授联合攻关的研究论文Regulation of phosphoribosyl ubiquitination by a calmodulin-dependent glutamylase。该研究发现致病性嗜肺军团菌(Legionella pneumophila)效应蛋白SidJ可以被宿主体内的一个钙调蛋白Calmodulin(简称CaM)结合并激活,进而通过谷氨酸化修饰(Glutamylation)抑制SidE家族泛素连接酶活性的分子机理【1】。该工作详细解析了致病性嗜肺军团菌效应蛋白SidJ调控SidE酶活性的分子机制,为探讨谷氨酸修饰酶(Glutamylase)的催化机制提供了新的体系,也为临床治疗嗜肺军团菌感染的药物开发提供了新靶点。

嗜肺军团菌是一种引起人的非典型性肺炎的条件性致病菌, 其通过IV型分泌系统Dot/Icm分泌300多个效应蛋白到宿主细胞中,调控干扰宿主各种信号通路以辅助其自身在宿主细胞内的增殖。对这些效应蛋白的功能解析不仅有助于阐明了嗜肺军团菌侵染宿主的分子机理,同时也能揭示新的细胞信号传导方式和蛋白质翻译后修饰模式【2】。众所周知,泛素化修饰几乎调控真核细胞的生命活动的所有方面【3】。在与宿主的长期博弈过程中,病原菌进化出了多种方式来干预宿主的泛素修饰系统,从而达到增强侵染效率和逃避宿主免疫监视的目的。2016年普渡大学罗招庆教授课题组在Nature杂志上首次报道了SidE家族效应蛋白独立于经典的E1和E2酶的泛素化修饰的重大发现【4】,引发了广泛关注【5-7】,该发现入选为当年度细胞信号传递领域的重大突破之一【8】。后续研究发现嗜肺军团菌中的另一个效应蛋白SidJ可调控SdeA等SidE家族成员(罗招庆组揭示独特的细菌去泛素化酶可逆转磷酸核糖连接的泛素化)【9】,但其生化机制尚未清楚。
针对这一重要科学问题,中美两国研究人员进行了联合攻关。首先,通过质谱(LC-MS/MS)分析,发现SidJ诱导失活的SdeA发生129.01道尔顿的分子量漂移。进一步研究发现,SidJ能对SdeA进行谷氨酸化修饰,且 SdeA 被修饰的位点是位于其mART结构域中860位的谷氨酸残基上,该位点是SdeA mART结构域催化的活性位点之一,主要负责水解NAD+,为泛素激活提供能量。更为有趣的是,研究者通过对SidJ序列分析,发现SidJ的C端具有一个结合CaM的保守IQ 结构域,CaM与SidJ结合形成稳定复合物,进而激活其酶活性。
随后研究人员解析了SidJ-CaM二元复合物以及SidJ-CaM结合AMP的三元复合物晶体结构,惊奇地发现SidJ的结构与在已知的真核生物中行使谷氨酸化修饰功能的tyrosineligase-like(TTLL)家族酶完全不同【10】。结构解析显示CaM能够通过N-lobe和C-lobe与SidJ存在广泛的结合。

SidJ-CaM(PDB:6K4K)以及SidJ-CaM-AMP(PDB:6K4R)的晶体结构及不同的ATP类似小分子对其激活的能力
另外,SidJ-CaM复合体结合一个AMP残基,揭示SidJ是在α位磷酸切割ATP,从而通过酰化单磷酸腺苷化激活被修饰位点的谷氨酸残基,异于常见的磷酸化激活。因此,g 位不能水解的类ATP分子仍能支持其活性。同时,在没有SdeA的反应体系里,SidJ能发生AMP自修饰(self-AMPylation),进一步说明该反应由两步特异的修饰完成。在没有受体基团的条件下,SidJ将源于ATP的AMP用于自修饰。综合现有的研究结果,我们提出了SidJ结合宿主CaM催化SdeA的谷氨酸化修饰,进而导致SdeA失活的分子机制(如下图)。
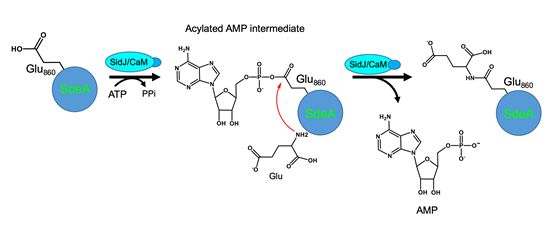
SidJ-CaM通过形成酰化单磷酸腺苷反应中间物催化谷氨酸化的反应模型。
谷氨酸化修饰此前多见于真核细胞微管蛋白(Tubulin)的修饰,并调控多种重要细胞功能,但是其催化机理如底物识别等机制并不是非常清楚【10,11】。SidJ作为一个CaM依赖的谷氨酸化酶为进一步探讨这一家族酶的作用机制提供了一个窗口。该工作是由美国普渡大学罗招庆教授课题组和福建师范大学欧阳松应教授课题组合作完成的。罗招庆教授和欧阳松应教授为本文的共同通讯作者,普渡大学罗招庆教授课题组博士生干宁海、福建师范大学生命科学学院青年教师甄向凯博士、普渡大学刘尧、以及福建师范大学南方生物医学研究中心一年级硕士生许小龙为共同第一作者。
原文链接:https://www.nature.com/articles/s41586-019-1439-1;
参考文献
1 Gan N, Zhen X, Liu Y, Xu X, He C, Qiu J, Liu Y, Fujimoto GM, Nakayasu ES, Zhou B, Zhao L, Puvar K, Das C, Ouyang S, Luo ZQ. Regulation of phosphoribosyl ubiquitination by a calmodulin-dependent glutamylase, Nature, doi:10.1038/s41586-019-1439-1 (2019).
2 Qiu,J. & Luo, Z. Q. Legionella and Coxiella effectors: strength in diversityand activity. Nat Rev Microbiol 15, 591-605,doi:10.1038/nrmicro.2017.67 (2017).
3 Komander,D. & Rape, M. The ubiquitin code. Annualreview of biochemistry 81,203-229, doi:10.1146/annurev-biochem-060310-170328 (2012).
4 Qiu,J., Sheedlo, M. J., Yu, K., Tan, Y., Nakayasu, E. S., Das, C., Liu, X. &Luo, Z. Q. Ubiquitination independent of E1 and E2 enzymes by bacterialeffectors. Nature 533, 120-124, doi:10.1038/nature17657(2016).
5 Bhogaraju,S. & Dikic, I. Cell biology: Ubiquitination without E1 and E2 enzymes. Nature 533, 43-44, doi:10.1038/nature17888 (2016).
6 Nakasone,M. A. & Huang, D. T. Ubiquitination Accomplished: E1 and E2 Enzymes WereNot Necessary. Mol Cell 62, 807-809,doi:10.1016/j.molcel.2016.06.001 (2016).
7 DuToit, A. Bacterial pathogenesis: Bacterial effectors skip a few steps. Nat Rev Microbiol 14, 331, doi:10.1038/nrmicro.2016.61 (2016).
8 Adler,E. M. Signaling Breakthroughs of the Year. Sci Signal 9, eg1,doi:10.1126/scisignal.aad9795 (2016).
9 Qiu,J., Yu, K., Fei, X., Liu, Y., Nakayasu, E. S., Piehowski, P. D., Shaw, J. B.,Puvar, K., Das, C., Liu, X. & Luo, Z. Q. A unique deubiquitinase thatdeconjugates phosphoribosyl-linked protein ubiquitination. Cell Res 27, 865-881,doi:10.1038/cr.2017.66 (2017).
10 Rogowski,K., van Dijk, J., Magiera, M. M., Bosc, C., Deloulme, J. C., Bosson, A., Peris,L., Gold, N. D., Lacroix, B., Bosch Grau, M., Bec, N., Larroque, C., Desagher,S., Holzer, M., Andrieux, A., Moutin, M. J. & Janke, C. A family ofprotein-deglutamylating enzymes associated with neurodegeneration. Cell 143, 564-578, doi:10.1016/j.cell.2010.10.014 (2010).
11 O'Hagan,R., Silva, M., Nguyen, K. C. Q., Zhang, W., Bellotti, S., Ramadan, Y. H., Hall,D. H. & Barr, M. M. Glutamylation Regulates Transport, SpecializesFunction, and Sculpts the Structure of Cilia. Curr Biol 27, 3430-3441e3436, doi:10.1016/j.cub.2017.09.066 (2017).
12 Bhogaraju S, Bonn F, Mukherjee R, Adams M, Pfleiderer MM, Galej WP, Matkovic V, Lopez-Mosqueda J, Kalayil S, Shin D, Dikic I. Inhibition of bacterial ubiquitin ligases by SidJ-calmodulin-catalysed glutamylation. Nature, doi: 10.1038/s41586-019-1440-8 (2019).
13 Black,M. H., Osinski, A., Gradowski, M., Servage, K. A., Pawlowski, K., Tomchick, D.R. & Tagliabracci, V. S. Bacterial pseudokinase catalyzes proteinpolyglutamylation to inhibit the SidE-family ubiquitin ligases. Science 364, 787-792, doi:10.1126/science.aaw7446 (2019).
专家 专家点评:朱永群(浙江大学生命科学研究院,教授)
2016年罗招庆教授课题组在Nature上发表文章,发现嗜肺军团菌SidE家族效应蛋白SdeA、SdeB、SdeC以及SidE能够以NAD为能量源,通过生成ADP-核糖基化(ADP-ribosylation)的泛素作为中间体,进行独特的蛋白质泛素化作用,从而首次提出E1和E2非依赖的蛋白质泛素化的全新概念。随后Ivan Dikic教授在他们工作的基础上,通过生化研究,以及Ralph Isberg教授通过细菌感染等实验,发现这种E1和E2非依赖的泛素化是SidE家族效应因子通过PDE(磷酸二酯酶)结构域,在其ADP核糖基转移酶结构域催化ADP-核糖基化泛素中间体的基础上,对激活的泛素(ADPR-Ub)进行切割,并将磷酸核糖基化的泛素共价连接到底物的丝氨酸残基上。这些研究改变了人们对蛋白质泛素化催化中三酶机制的传统观念。
然而在SidE家族基因座中间存在一个SidJ基因。2007年罗招庆教授首先克隆出SidJ基因,并发现其对嗜肺军团菌的胞内生存具有重要作用。随后在2015年,Craig Roy教授以及Joseph Vogel教授都发现了SidJ能够抑制SidE家族效应蛋白对酵母细胞的毒性作用,表明细菌本身存在对这些泛素连接酶功能的调节,然而其中的机制并不清楚。在这项研究中,罗招庆教授课题组首先通过酵母毒性实验,发现SidJ抑制SdeA的ADP-核糖基转移酶酶的活性,然后通过质谱测定,发现SidJ是一个独特的谷氨酸化酶,对SdeA的ADP-核糖基化转移酶结构域中的活性氨基酸残基E860进行了一种不常见的谷氨酸化修饰,修饰后的SdeA丧失了ADP-核糖基化转移酶活性,故而不能进行随后的E1和E2非依赖的泛素化反应。他们早在2011年时就通过对SidJ序列分析发现SidJ能够结合真核生物所特有的钙调蛋白,但其结合的生物学意义一直不清楚。在确认SidJ可能谷氨酸化修饰SdeA后,他们通过生化实验,发现钙调蛋白的结合激活了SidJ的谷氨酸化酶活性。通过与福建师范大学的欧阳松应教授合作,成功解析了SidJ与钙调蛋白以及AMP的不同复合物的晶体结构,发现SidJ可能通过腺苷单磷酸化作用对SdeA进行谷氨酸化修饰的全新酶学机理。
这项研究首次发现病原菌效应蛋白具有独特的谷氨酸化酶学活性,揭示了病原菌利用本身蛋白和真核生物特有分子对细菌的感染过程进行精确调节的新机制,也促进了人们对多见于对真核细胞微管蛋白修饰的谷氨酸化修饰的催化机制的新认识。另外,Ivan Dikic教授直接通过真核共表达和质谱鉴定的方法,以及西南医学中心的Vincent S. Tagliabracci助理教授从生物信息学、分析SidJ假激酶结构域的角度入手,获得了相似的发现,说明了多学科交叉非常有利于从不同角度去全面地认识同一生物学现象及其分子机制。
(转载自BioART)
2. 欧阳松应教授课题组又一CRISPR-Cas系统结构与功能方面最新进展研究成果在《Nature》子刊上发表
2019年6月11日,欧阳松应教授课题组在CRISPR-Cas系统结构与功能方面取得最新进展,研究成果以题为“Two HEPN domains dictate CRISPR RNA maturation and target cleavage in Cas13d”发表在《Nature》子刊《Nature Communications》((2019) 10:2544, https://doi.org/10.1038/s41467-019-10507-3)(影响因子:12.353)。生命科学学院为第一完成单位,欧阳松应教授为独立通讯作者。这是该课题组在《Cell Research》(2018,影响因子:15.353)期刊上发表该领域研究成果之后的又一力作[2]。该研究解析了瘤胃球菌uncultured Ruminococcus sp.Cas13d(简称UrCas13d)蛋白与CRISPR RNA(简称crRNA)二元复合物高分辨率晶体结构,揭示了UrCas13d加工precursor crRNA(简称pre-crRNA)的酶活性关键位点,UrCas13d对crRNA repeat region的核酸序列与结构的特异性识别,两个水合镁离子对于稳定crRNA repeat region的构象发挥着重要作用,鉴定了crRNA spacer region和靶RNA之间的错配耐受性等尚未研究清楚的科学问题。本研究成果为进一步改造Cas13d作为核酸编辑及检测工具,并发挥其在解决人类疾病与健康等问题上的应用潜力,提供了扎实的结构基础和理论依据。

CRISPR-Cas系统是原核生物中存在一种规律成簇的间隔短回文重复DNA序列。CRISPR-Cas系统表达的非编码RNA(CRISPR RNA,crRNA)及其相关蛋白(CRISPR associated protein, Cas)组成的复合物使得原核生物能够识别并且抵御外源核酸物质的入侵。CRISPR-Cas系统主要分为两类,它们各自又由多个类型及亚型组成。第二大类CRISPR-Cas系统中的II型CRISPR-Cas9系统和V型CRISPR-Cas12a(Cpf1)系统,已经广泛并高效地应用于多种生物的基因组编辑。Cas9可以纠正个体细胞内的基因缺陷,已经从实验室研究阶段进入到临床研究阶段,具有治愈或预防多种人类疾病的潜力;然而,Cas9系统和Cas12a系统主要负责编辑DNA。针对RNA的编辑工具可以在RNA水平调节基因的功能,而不会对基因本身造成永久性改变。
2018年4月,美国Salk研究所Patrick Hsu课题组和Arbor生物技术公司分别报道了一种新型Cas效应蛋白,属于第二大类VI型RNA依赖的核糖核酸酶(RNase),并命名为Cas13d,该蛋白可做为一种强大RNA编辑工具用于纠正痴呆症患者细胞中引起疾病的蛋白质失衡[3, 4]。与VI型其他成员类似,Cas13d具有两个HEPN(higher eukaryotes and prokaryotes nucleotide-binding)结构域,自身可以加工pre-crRNA成为成熟的crRNA,结合靶RNA激活其RNase活性。虽然,Cas13d在功能上与其家族成员Cas13a-c具有一定的相似性,但是Cas13d具备一些独特之处。首先,Cas13d的蛋白序列平均长度为930个氨基酸残基,在同类蛋白中明显偏小(比其家族成员Cas13a-c小20%,比Cas9小33%),使它更容易包装到容量有限的应用载体中(比如adeno-associated virus, AAV);其次,除了两个HEPN结构域之外,Cas13d与其他Cas13家族成员之间没有明显的序列相似性;另外,在切割靶RNA时,Cas13d的靶RNA序列两端都无须PFS(protospacer flanking sequence);此外,含有WYL结构域的辅助蛋白可以增强Cas13d的底物酶切活性。因此,Cas13d更有潜力作为一种可操控的RNA结合模块,有效靶向细胞内的RNA转录物,为转录组工程和疾病治疗提供了一个通用平台。2018年9月,美国Salk研究所Patrick Hsu课题组和Dmitry Lyumkis课题组合作,利用低温电镜技术率先报道了惰性真杆菌Eubacterium siraeum Cas13d(EsCas13d)不同功能状态下的复合物冷冻电镜结构[5]。
本研究中,欧阳松应教授课题组解析了UrCas13d蛋白和crRNA二元复合物1.86Å高分辨率的晶体结构,并开展了相应的功能实验[1]。发现在UrCas13d-crRNA二元复合物中,成熟的crRNA结合在UrCas13d的紧凑双叶型结构所形成的通道内,两个水合镁离子对于稳定crRNA repeat region的构象发挥着重要作用;其中一个五水合镁离子通过多个氢键稳定crRNA repeat region的构象,将与该水合镁离子直接相互作用的氨基酸残基突变,发现靶RNA切割活性显著降低。进一步的实验表明,二价金属离子对于pre-crRNA加工不是必需的,但对于Cas13d的靶RNA切割是至关重要的;与Cas13家族其他成员的研究结果相比所不同的是,UrCas13d蛋白加工pre-crRNA的酶活性中心位于HEPN-2结构域上,其氨基酸残基R802和K905对于pre-crRNA切割是极其重要的;crRNA repeat region的核苷酸U(-8)-C(-1)的序列和结构被UrCas13d特异性识别,该区域的核酸序列和三维结构对于靶RNA的切割是必需的;在二元复合物中,crRNA repeat region的核苷酸U(-24)-A(-13)暴露于环境溶剂中,删除该区域内四个Watson-Crick碱基对(base pair)既不改变UrCas13d的pre-crRNA加工能力也不改变其靶RNA切割能力;针对UrCas13d两个HEPN结构域上靶RNA切割关键位点R288A或H828A,突变体蛋白足以破坏蛋白对靶RNA切割活性。此外,通过crRNA spacer region和靶RNA之间的错配耐受性实验,研究发现spacer region的两个子区域(an internal and a 3-end region)与靶RNA之间的正确碱基配对对于靶RNA切割是必需的。
我院副研究员张博博士和研究生叶阳苗为本文的共同第一作者,该研究得到国家自然科学基金和福建师范大学的经费资助,上海同步辐射光源(SSRF)BL-17U1对该研究提供了重要的技术支持。
原文链接:https://www.nature.com/articles/s41467-019-10507-3
参考文献
[1].Zhang, B. et al. Two HEPN domains dictate CRISPR RNA maturation and target cleavage in Cas13d. Nature Communications. 10:2544 (2019). DOI:10.1038/s41467-019-10507-3.
[2].Zhang, B. et al. Structural insights into Cas13b-guided CRISPR RNA maturation and recognition. Cell Res28, 1198–1201 (2018).
[3].Konermann, S. et al. Transcriptome Engineering with RNA-Targeting Type VI-D CRISPR Effectors. Cell173, 665-676 e14 (2018).
[4].Yan, W.X. et al. Cas13d Is a Compact RNA-Targeting Type VI CRISPR Effector Positively Modulated by a WYL-Domain-Containing Accessory Protein. Mol Cell70, 327-339 e5 (2018).
[5].Zhang, C. et al. Structural Basis for the RNA-Guided Ribonuclease Activity of CRISPR-Cas13d. Cell175, 212-223 e17 (2018).
3. Cas13b介导的CRISPR RNA成熟和识别的结构机制
2018年11月13日,Nature旗下Cell Research杂志在线发表了本课题组最新研究成果,文章标题为“Structural insights into Cas13b-guided CRISPR RNA maturation and recognition”。

该研究解析了Bergeyella zoohelcum(动物溃疡伯格菌)中Cas13b(BzCas13b)与CRISPR RNA (crRNA) 二元复合物的晶体结构,揭示了precursor crRNA(pre-crRNA)成熟加工的酶活位点,阐明了crRNA成熟的分子机制;该研究成果对认识与理解细菌抵抗RNA病毒入侵的分子基础具有重要的意义,同时也为改造CRISPR-Cas13b系统,为其在RNA识别与编辑、核酸检测领域的应用提供了坚实的结构生物学基础。
本研究中,BzCas13b蛋白全长由1224个氨基酸残基构成,它包括了多个结构域。在课题组解析的其与crRNA二元复合物晶体结构中(分辨率为2.79 Å),他们发现BzCas13b的多结构域以L型crRNA为中心环绕分布,整体呈现出三角形空间结构,其Helical-1, HEPN-1 and HEPN-2结构域构成三角形结构的一边,RRI-1、RRI-2结构域和linker region一起构成三角形结构的另一边,Helical-2结构域构成三角形结构的第三边。成熟的crRNA也包括了4个亚区域。结合结构生物学和体外生物化学等实验手段,课题组首次揭示了加工pre-crRNA的蛋白酶活位点,发现了pre-crRNA被加工的重要位点,校正了以往相关研究领域中不准确的研究内容,并且进一步阐明了crRNA的识别机制。另外,之前的相关研究表明,来自BzCas13b的两个HEPN结构域的氨基酸残基R116,H121,R1177和H1182参与了靶RNA的切割,本研究内容证明了单个R116A或R1177突变足以破坏该蛋白的靶RNA切割活性。
欧阳松应教授为本文的独立通讯作者,欧阳松应教授课题组的青年教师张博博士和研究生叶维伟为本文的共同第一作者,该研究得到国家自然科学基金和福建师范大学的经费资助,上海同步辐射光源(SSRF)BL-17U1对该研究提供了重要的技术支持。
据悉,Cell Research杂志是Nature旗下一个同行评议的国际期刊(月刊),发表生物-细胞生物学领域最新研究进展的原创性研究论文、权威综述、快报和述评的国际性期刊,最新影响因子:15.393,I区。
论文链接:https://www.nature.com/articles/s41422-018-0109-4
五、代表性论文
1. Fu J#, Li P#, Guan H#, Huang D, Song L*, Ouyang S*, Luo ZQ*. Legionella pneumophila temporally regulates the activity of ADP/ATP translocases by reversible ADP-ribosylation. mLife. 2022. Accepted. (*共同通讯作者)
2. Zhang B#, Luo D#, Li Y#, Perculija V, Chen J, Lin J, Ye Y, Ouyang S*. Mechanistic insights into the R-loop formation and cleavage in CRISPR-Cas12i1. Nat Commun. 2021 Jun 9;12(1):3476. (*独立通讯作者)
3. Perčulija V, Lin J, Zhang B*, Ouyang S*. Functional features and current applications of the RNA-targeting type VI CRISPR-Cas systems. Advanced Science. 2021 May 5;8(13):2004685. DOI: 10.1002/advs.202004685 (*共同通讯作者)
4. Zhang S#, Xu Y#, Guan H#, Cui T#, Liao Y#, Wei W#, Li J, Hassan BH, Zhang H, Jia X, Ouyang S*, Feng Y*. Biochemical and structural characterization of the BioZ enzyme engaged in bacterial biotin synthesis pathway. Nat Commun. 2021 Apr 6;12(1):2056. doi: 10.1038/s41467-021-22360-4. (*共同通讯作者)
5. Xu X#, Zhang D#, Zhou B, Zhen X*, Ouyang S*. Structural and biochemical analysis of the tetrameric cell binding domain of Lys170 from enterococcal phage F170/08. Eur Biophys J. 2021 Feb 20. doi: 10.1007/s00249-021-01511-x. (*共同通讯作者)
6. Saeed AFUH, Su J*, Ouyang S*. Marine-derived drugs: Recent advances in cancer therapy and immune signaling. Biomedicine & Pharmacotherapy. 2020 Dec 16;134:111091. doi: 10.1016/j.biopha.2020.111091. (*共同通讯作者)
7. Liu S#, Luo J#, Zhen X, Qiu J*, Ouyang S* and Luo ZQ*. Interplay between bacterial deubiquitinase and ubiquitin E3 ligase regulates ubiquitin dynamics on Legionella phagosomes. eLife. 2020 Nov 2;9:e58114. doi: 10.7554/eLife.58114. (*共同通讯作者)
8. Yao J#, Zhen X#, Tang K, Liu T, Xu X, Chen Z, Guo Y, Liu X, Wood TK, Ouyang S*, Wang X*. Novel polyadenylylation-dependent neutralization mechanism of the HEPN/MNT toxin/antitoxin system. Nucleic Acids Res. 2020 Nov 4;48(19):11054-11067. (*共同通讯作者)
9. Guan H#, Wang Y#, Perčulija V, Saeed AFUH, Liu Y, Li J, Jan SS, Li Y, Zhu P*, Ouyang S*. Cryo-electron Microscopy Structure of the Swine Acute Diarrhea Syndrome Coronavirus Spike Glycoprotein Provides Insights into Evolution of Unique Coronavirus Spike Proteins. J Virol. 2020 Aug 19:JVI.01301-20. doi: 10.1128/JVI.01301-20. (*共同通讯作者)
10. Wang ZX, Liu SB, Guan H, Lu LF, Li S, Tu JG, Ouyang S*, Zhang YA*. Structural and Functional Characterization of Spring Viremia of Carp Virus Phosphoprotein Oligomerization Domain. J Virol. 2020 Jul 16;94(15):e00855-20. doi: 10.1128/JVI.00855-20 (*共同通讯作者)
11. Saeed AF, Ouyang S*. Ubiquitin: There's no quitting. Science Bulletin. DOI: 10.1016/j.scib.2020.04.026. (*独立通讯作者,Research Highlights)
12. Guan H#, Fu J#, Yu T#, Wang ZX#, Gan N, Huang Y, Perčulija V, Li Y, Luo ZQ*, Ouyang S*. Structural basis of ubiquitination catalyzed by the bacterial transglutaminase MavC. Advanced Science. 2020. DOI: 10.1002/advs.202000871. (*共同通讯作者)
13. Zhou B#, Zhen X#, Zhou H#, Zhao F#, Fan C, Perčulija V, Tong Y, Mi Z*, Ouyang S*. Structural and functional insights into a novel two-component endolysin encoded by a single gene in Enterococcus faecalis phage. PLoS Pathogens. 2020 Mar 16; 16 (3), e1008394. DOI: 10.1371/journal.ppat.1008394. (*共同通讯作者)
14. Guan H#, Wang Y#, Yu T#, Huang Y#, Li M, Saeed AFUH, Perčulija V, Li D, Xiao J, Wang D, Zhu P*, Ouyang S*. Cryo-EM structures of the human PA200 and PA200-20S complex reveal regulation of proteasome gate opening and two PA200 apertures. PLoS Biol. 2020 Mar 5;18(3):e3000654. doi: 10.1371/journal.pbio.3000654. (*共同通讯作者,杂志网站头条推荐)
15. Saeed A#, Ruan X#, Guan H, Su J, Ouyang S*. Regulation of cGAS-mediated Immune Responses and Immunotherapy. Advanced Science. 2020, 7, 1902599. DOI:10.1002/advs.201902599. (*独立通讯作者)
16. Ma K#, Zhen X#, Zhou B#, Gan N, Fan C, Ouyang S*, Luo Z*, Qiu J*. Regulation of the association of K63-linked polyubiquitins on Legionella phagosome by the deubiquitinase Ceg23. Journal of Biological Chemistry. 2020 Feb 7;295 (6), 1646-1657 (*共同通讯作者)
17. Wang ZX, Lu LF, Lu XB, Ni B, Liu MX, Guan H, Li S, Zhang YA*, Ouyang S*. Infectious Hematopoietic Necrosis Virus N Protein Suppresses Fish IFN1 Production by Targeting the MITA. Fish & Shellfish Immunology. 2019 Dec 24;97:523-530. (*共同通讯作者,I区)
18. Gan N#, Guan H#, Huang Y, Yu T, Nakayasu E, Puvar K, Das C, Wang D, Ouyang S*, Luo ZQ*. Legionella pneumophila regulates the activity of the E2 ubiquitin conjugating enzyme UBE2N by deamidase-mediated deubiquitination. EMBO J. 2019 Dec 11: 39 (4)e102806. doi: 10.15252/embj.2019102806. (*共同通讯作者,影响因子:11.227)
· News & Views: Yan F, Zhu Y, Zhou Y*. Legionella pneumophila balances ubiquitin transglutamination. EMBO J. 2020 Jan 28:e104258. DOI: 10.15252/embj.2019104258
19. Wei W#, Guan H#, Zhu T#, Zhang S#, Fan C, Ouyang S*, Feng Y*. Molecular Basis of BioJ, a Unique Gatekeeper in Bacterial Biotin Synthesis. iScience. 2019 Aug 22;19:796-808. (*共同通讯作者,Cell子刊)
20. Gan N#, Zhen X#, Liu Y#, Xu X#, He C, Qiu J, Liu Y, Fujimoto GM, Nakayasu ES, Zhou B, Zhao L, Puvar K, Das C, Ouyang S*, Luo ZQ*. Regulation of phosphoribosyl ubiquitination by a calmodulin-dependent glutamylase. Nature. DOI: 10.1038/s41586-019-1439-1. (*共同通讯作者)
Comment in:Glutamylation of Bacterial Ubiquitin Ligases by a Legionella Pseudokinase. Sulpizio AG, Minelli ME, Mao Y. Trends Microbiol. 2019 Dec;27(12):967-969. doi: 10.1016/j.tim.2019.09.001. Epub 2019 Oct 14.
21. Song X#, Shan H#, Zhu Y#, Hu S, Xue L, Chen Y, Ding W, Niu T, Gu J, Ouyang S*, Shen QT*, Liu ZJ*. Self-capping of nucleoprotein filaments protects Newcastle Disease Virus genome. elife. 2019 Jul 10;8. pii: e45057. doi: 10.7554/eLife.45057. (*共同通讯作者)
22. Zhang B#, Ye Y#, Ye W, Perčulija V, Jiang H, Chen Y, Li Y, Chen J, Lin J, Wang S, Chen Q, Han YS, Ouyang S*. Two HEPN domains dictate CRISPR RNA maturation and target cleavage in Cas13d. Nat Commun. 2019 Jun 11;10(1):2544. doi: 10.1038/s41467-019-10507-3. (*独立通讯作者)
23. Wu S#, Zhang Q#, Zhang F#, Meng F, Liu S, Zhou R, Wu Q, Li X, Shen L, Huang J, Qin J, Ouyang S, Xia Z, Song H, Feng XH, Zou J, Xu P*. HER2 recruits AKT1 to disrupt STING signalling and suppress antiviral defense and antitumor immunity. Nat Cell Biol. 2019 Jul 22. doi: 10.1038/s41556-019-0352-z.
24. Yang P#, Huang X#, Lai C#, Li L#, Li T, Huang P, Ouyang S, Yan J, Cheng S, Lei G, Wang Z, Yu L, Hong Z, Li R, Dong H, Wang C, Yu Y, Wang X, Li X, Wang L, Lv F, Yin Y, Yang H, Song J, Gao Q*, Wang X*, Zhang S*. SET domain containing 1B gene (SETD1B) is mutated in primary hepatic neuroendocrine tumors. Int J Cancer. 2019 Apr 11. doi: 10.1002/ijc.32334.
25. Zhang B#, Ye W#, Ye Y, Zhou H, Saeed AFUH, Chen J, Lin J, Perčulija V, Chen Q, Chen CJ, Chang MX, Choudhary MI, Ouyang S*. Structural insights into Cas13b-guided CRISPR RNA maturation and recognition. Cell Res. 2018 Nov 13. doi: 10.1038/s41422-018-0109-4. (*独立通讯作者)
26. Zhen X, Zhou H, Ding W, Zhou B, Xu X, Perčulija V, Chen CJ, Chang MX, Choudhary MI, Ouyang S*. Structural basis of pheromone recognition by AimR among Spbeta group bacteriophages. Protein Cell. 2018 Nov 12. doi: 10.1007/s13238-018-0588-6. (*独立通讯作者)
27. Qu L, Jiang Y, Cheng C, Wu D, Meng B, Chen Z, Zhu Y, Shaw N, Ouyang S*, Liu ZJ*. Crystal structure of ATP-bound human ABCF1 demonstrates a new conformation of ABC proteins. Structure. 2018 Sep 4;26(9):1259-1265.e3. (*共同通讯作者)
28. Li J#, Rodriguez J#, Niu F, Pu M, Wang J, Hung LW, Shao Q, Zhu Y, Ding W, Liu Y, Da Y, Yao Z, Yang J, Zhao Y, Wei GH, Cheng G, Liu ZJ, Ouyang S*. Structural basis for DNA recognition by STAT6. Proc Natl Acad Sci USA. 2016 Nov 15;113(46):13015-13020. (*独立通讯作者。This article is a PNAS Direct Submission)
· 同行评价:1). 王璞玥*, 杨正宗, 杜生明. STAT6识别N4位点DNA的结构基础被揭示. SCIENTIA SINICA Vitae. 2017 Jan; 47(2), 240-243.
2). Zhou X, Jiang Z*. N4 DNA Recognition by STAT6 – Structural and Functional Implications. Protein Cell. 2017 Apr;8(4):240-241.
29. Jiang Y, Zhu Y, Liu ZJ, Ouyang S*. The emerging roles of the DDX41 protein in immunity and diseases. Protein Cell. 2017 Feb;8(2):83-89. (*独立通讯作者)
30. Jiang Y, Zhu Y, Qiu W, Liu YJ, Cheng G, Liu ZJ*, Ouyang S*. Structural and functional analyses of human DDX41 DEAD domain. Protein Cell. 2017 Jan;8(1):72-76. (*共同通讯作者)
31. Chu Y#, Zhu Y#, Chen Y, Li W, Zhang Z, Liu D, Wang T, Ma J, Deng H, Liu ZJ, Ouyang S*, Huang L*. aKMT Catalyzes Extensive Protein Lysine Methylation in the Hyperthermophilic Archaeon Sulfolobus islandicus but is Dispensable for the Growth of the Organism. Mol Cell Proteomics. 2016 Sep;15(9):2908-23. (*共同通讯作者)
32. Wang L#, Valderramos SG#, Wu A, Ouyang S, Li C, Brasil P, Bonaldo M, Coates T, Nielsen-Saines K, Jiang T*, Aliyari R*, Cheng G*. From Mosquitos to Humans: Genetic Evolution of Zika Virus. Cell Host Microbe. 2016 Apr 14. pii: S1931-3128(16)30142-1. (已被引用50次)
33. Jiang Y, Jiang H, Zhou S, Meng B, Liu ZJ*, Ouyang S*. Crystal structure of hGEF-H1 PH domain provides insight into incapability in phosphoinositide binding. Biochem Biophys Res Commun. 2016 Mar 18;471(4):621-7. (*共同通讯作者)
34. Ni X#, Ru H#, Ma F#, Zhao L#, Shaw N, Feng Y, Ding W, Gong W, Wang Q, Ouyang S*, Cheng G*, Liu ZJ*. New insights into the structural basis of DNA recognition by HINa and HINb domain of IFI16. J Mol Cell Biol. 2016 Jan 12;8 (1): 51-61. (*共同通讯作者,封面文章)
35. Liu B#, Ouyang S#, Makarova K, Xia Q, Zhu Y, Li Z, Guo L, Koonin E, Liu ZJ*, Huang L*. A primase subunit essential for efficient primer synthesis by an archaeal eukaryotic-type primase. Nat Commun. 2015 Jun 22;6:7300. (#共同第一作者)
36. Zhao L#, Hua T#, Crowley C, Ru H, Ni X, Shaw N, Jiao L, Ding W, Jiang Y, Hung LW, Liu L, Ye K, Ouyang S*, Cheng G*, Liu ZJ*. Structural analysis of asparaginyl endopeptidase reveals the activation mechanism and a reversible intermediate maturation stage. Cell Res. 2014 Mar; 24(3): 344-58. (*共同通讯作者,已被引用25次)
37. Gu J, Feng Y, Feng X, Sun C, Lei L, Ding W, Niu F, Jiao L, Yang M, Li Y, Liu X, Song J, Cui Z, Han D, Du C, Yang Y, Ouyang S*, Liu ZJ*, Han W*. Structural and biochemical characterization reveals LysGH15 as an unprecedented “EF-hand-like” calcium-binding phage lysin. PLOS Pathogens. 2014 May 15;10(5):e1004109. (*共同通讯作者,已被引用16次)
38. Meng B#, Wu D#*, Gu J, Ouyang S, Ding W, Liu ZJ*. Structural and functional analyses of human tryptophan 2,3-dioxygenase. 2014. PROTEINS: Structure, Function, and Bioinformatics. 2014 Nov;82(11):3210-6.
39. Jiao L#, Ouyang S#, Shaw N, Song G, Feng Y, Niu F, Qiu W, Zhu H, Hung LW, Zuo X, Shtykova E, Zhu P, Dong YH, Xu R*, Liu ZJ*. Mechanism of the Rpn13-induced activation of Uch37. Protein Cell. 2014 Aug;5(8):616-30. (#共同第一作者)
40. Zhang L, Mo J, Swanson K, Wen H, Petrucelli A, Gregory S, Zhang Z, Schneider M, Jiang Y, Fitzgerald K, Ouyang S, Liu ZJ, Damania B, Shu HB, Duncan J, Ting J*. NLRC3, a member of the NLR family of proteins, is a negative regulator of innate immune signaling induced by the DNA sensor STING. Immunity. 2014 Mar 20; 40(3): 329-341. (已被引用69次)
· 同行评价:Mangan M, Latz E*. NLRC3 Puts the Brakes on STING. Immunity. 2014 Mar 20; 40(3): 305-306.
41. Niu F, Ru H, Ding W, Ouyang S*, Liu ZJ*. Structural biology study of human TNF receptor associated factor 4 TRAF domain. Protein Cell. 2013, 4(9): 687-694. (*共同通讯作者,封面文章)
42. Niu F#, Shaw N#, Wang Y#, Jiao L#, Ding W, Upurd H, Ouyang S*, Cheng G*, Liu ZJ*. From the Cover: Structure of the Leanyer Orthobunyavirus Nucleoprotein-RNA complex reveals novel architecture for RNA encapsidation. Proc Natl Acad Sci USA. 2013 May 28;110(22):9054-9. (*共同通讯作者,已被引用30次。This article is a PNAS Direct Submission)
· 同行评价:Zheng W, Tao YJ*. Genome encapsidation by orthobunyavirus nucleoproteins. Proc Natl Acad Sci U S A. 2013 110 (22) 8769-8770.
43. Jiao L#, Ouyang S#, Liang M#, Niu F#, Shaw N, Wu W, Ding W, Jin C, Peng Y, Zhu Y, Zhang F, Wang T, Li C, Zuo X, Luan CH, Li D, Liu ZJ*. Structure of severe fever with thrombocytopenia syndrome virus nucleoprotein in complex with Suramin reveals therapeutic potentials. J Virol. 2013 Jun;87(12):6829-39. (#共同第一作者,已被引用38次)
44. Shaw N, Ouyang S, Liu ZJ*. Binding of bacterial secondary messenger molecule c di-GMP is a STING operation. Protein Cell. 2013 Feb;4(2):117-29.
45. Parvatiyar K#, Zhang Z#, Teles RM, Ouyang S, Jiang Y, Iyer SS, Zaver SA, Schenk M, Zeng S, Zhong W, Liu ZJ, Modlin RL, Liu YJ*, Cheng G*. The helicase DDX41 recognizes the bacterial secondary messengers cyclic di-GMP and cyclic di-AMP to activate a type I interferon immune response. Nat Immunol. 2012 Nov 11; 13(8): 1155-1161. (封面文章, 已被引用达187次)
· 同行评价:“News and Views”by Andrew G Bowie:Innate sensing of bacterial cyclic dinucleotides: more than just STING. Nat Immunol. 2012 Dec;13(12):1137-9.
46. Ouyang S#, Song X#, Wang Y#, Ru H#, Shaw N, Jiang Y, Niu F, Zhu Y, Qiu W, Parvatiyar K, Li Y, Zhang R, Cheng G*, Liu ZJ*. Structural Analysis of the STING Adaptor Protein Reveals a Hydrophobic Dimer Interface and Mode of Cyclic di-GMP Binding. Immunity. 2012 Jun; 36(6): 1073-1086. (#共同第一作者,已被引用达128次) (作为当年具有重要影响力的科研成果被中国科学院《2013科学发展报告》收录为“中国科学家代表性成果”;被《中国科学院重大成果年报(2012)》收录,入选《中国科学院国际科技合作年度述评》2012年度报告的《国际科技合作重要进展》)
47. Ouyang S#, Gong B#, Li JZ#, Zhao L#, Wu W, Zhang FS, Sun L, Wang SJ, Pan M, Li C, Liang W, Shaw N, Zheng J, Zhao GP, Wang Y*, Liu ZJ*, Liang M*. Structural insights into a human anti-IFN antibody exerting therapeutic potential for systemic lupus erythematosus. J Mol Med (Berl). 2012 July; 90(7):837-846. (#共同第一作者)
48. Zhan Z#, Ouyang S#, Liang W, Zhang Z, Liu ZJ*, Huang L*. Structural and functional characterization of the C-terminal catalytic domain of the SSV1 integrase. Acta Crystallogr D Biol Crystallogr. 2012 Jun; 68(Pt 6):659-70. (#共同第一作者)
49. Liang W#, Ouyang S#, Shaw N, Joachimiak A, Zhang R, Liu ZJ*. Conversion of D-ribulose 5-phosphate to D-xylulose 5-phosphate: new insights from structural and biochemical studies on human RPE. FASEB J. 2011 Feb; 25(2):497-504.(#共同第一作者)
50. Qiu XB#*, Ouyang S#, Li CJ, Miao S, Wang L, Goldberg AL*. hRpn13/ADRM1/GP110 is a novel proteasome subunit that binds the deubiquitinating enzyme, UCH37. EMBO J. 2006 Dec 13;25(24):5742-53. (#共同第一作者,已被引用达158次)
51. Yin XQ#, Ouyang S#, Xu WM*, Zhang XP, Fok K.L., Zhang JP, Miao SY, Chan HC*, Wang LF*. YWK-II as a novel Go protein-coupled receptor for Müllerian Inhibiting Substance in cell survival. J Cell Sci. 2007 May 1;120(Pt 9):1521-8. (#共同第一作者)
六、主要出版论著
汪少芸,欧阳松应,主编《生物分子分离与表征》,科学出版社, ISBN:978-7-03-070082-7,2021年11月出版。
甄向凯,欧阳松应。参编《噬菌体学:从理论到实践》第29章《噬菌体冷冻电子显微镜技术》,科学出版社,ISBN:978-7-03-069286-3,2021年9月出版。
Perčulija V, Ouyang S*. Diverse Roles of DEAD/DEAH-Box Helicases in Innate Immunity and Diseases. A chapter to HELICASES FROM ALL DOMAINS OF LIFE, edited by Dr. RENU TUTEJA, ICGEB, New Delhi. ISBN: 9780128146866. Elsevier出版社. 2018年9月.(*通讯作者)
欧阳松应,刘志杰。重要天然免疫系统信号分子STING的结构与功能研究取得重要进展.《2013科学发展报告》,科学出版社,ISBN: 978-7-03-036755-6/N.497
参编《生物信息学方法指南》一书(科学出版社,ISBN 7-03-014465-1,2005年2月)。